早大など、進化の過程で失われた可能性がある幻のタンパク質構造を発見
マイナビニュース / 2024年7月23日 19時11分
早稲田大学(早大)と理化学研究所(理研)の両者は7月19日、進化のミッシングリンクとなる新しいタンパク質構造を発見し、これを用いることで、遺伝子発現系に重要なタンパク質構造の進化を実験的に再現することに成功したと共同で発表した。
同成果は、早大 人間科学学術院の八木創太講師(理研 生命機能科学研究センター(BDR) 客員研究員兼任)、BDRの田上俊輔チームリーダーらの共同研究チームによるもの。詳細は、英オンライン科学誌「Nature Communications」に掲載された。
細胞内の遺伝子発現機構では、DNAから転写反応によりRNAが作られ、RNAの翻訳反応によってタンパク質が作り出される。これまで、同機構の詳細な制御の仕組みについて数多くの研究が行われてきたが、太古の地球上で同機構がどのようにして成立してきたのかについては、まだ多くの謎が残されているという。
転写反応ではタンパク質の一種の「RNAポリメラーゼ」が、翻訳反応では、RNA・タンパク質複合体の「リボソーム」がそれぞれ重要な役割を担う。それらは巨大な構造を持つが、初期の生命が最初からそのような複雑な分子を持っていたとは考えにくく、当時はもっと小さくて単純な分子だったと推定されている。
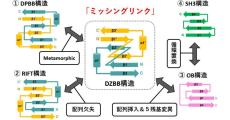
両者の共通項は、小型の「βバレル型構造」を重要な構成要素として有している点だ。たとえば、RNAポリメラーゼの活性コアを形成する「Double-psiβ-barrel(DPBB)構造」、リボソーム「L3」が持つ「RIFT構造」、複数のリボソームに高度に保存される「OBフォールド」や「SH3フォールド」などが挙げられる。これらの構造は部分的な類似点も多いことから、その進化的関係性についての議論がなされていたが、これらのβバレル構造間のアミノ酸配列の違いは大きく、実際どのようにして多様なβバレル構造が進化してきたのかは未解明だったとする。
そうした中で早大の八木講師らは、DPBB構造が7種類のアミノ酸を43個をつなげた単純なペプチドで再構成できることを実証。この単純なペプチドは水溶液中では特定の構造を形成できないが、マロン酸やリンゴ酸などの有機酸を含む溶液中では結晶化してDPBB構造を作ることがわかっていた。そこで研究チームは今回、この単純なペプチドが硫酸イオンを含む条件でも結晶を形成するのか調べしたという。
すると、単純なペプチドが硫酸イオンを含む条件でも結晶を形成し、DPBBとは異なるβバレル構造を作ることが見出された。この構造は、これまで自然界では未発見の新たな構造であり、立体構造を模式的に表すとギリシャ文字のZに似た特徴的なループ構造を2つ持つことから、「Double-zetaβ-barrel(DZBB)構造」と命名された。つまり、この単純なペプチドは同じ配列でも異なる条件では異なるタンパク質構造を作るメタモルフィック(変性作用のある)タンパク質であることが判明したのである。
-
-
- 1
- 2
-
この記事に関連するニュース
-
新しいタンパク質構造を発見
共同通信PRワイヤー / 2024年7月19日 11時0分
-
「プログラム可能」な新DNA編集技術 東大などが発表 さまざまな2つのDNAを切って組み換え
ITmedia NEWS / 2024年7月11日 8時0分
-
2型自然免疫細胞(ILC2)の新規分化メカニズムを解明~難治性アレルギー疾患の新規治療法開発に期待~
PR TIMES / 2024年7月9日 11時45分
-
トランスファーRNAを修飾する酵素が脳機能を支えるしくみを解明− RNA修飾の欠失による脳の病気の一般原理の理解へ −
PR TIMES / 2024年7月2日 12時45分
-
アレルギー疾患に関連するゲノム領域の重要性を解明~気管支ぜんそくの新規治療法の開発に期待~
PR TIMES / 2024年6月27日 10時45分
ランキング
-
1知って納得、ケータイ業界の"なぜ" 第173回 総務省が打ち出す1万5000円の値引き緩和ではミリ波対応スマホが到底普及しない理由
マイナビニュース / 2024年7月23日 17時56分
-
2「週刊文春」記者かたる“なりすましメール”相次ぐ 著名人に嫌がらせ&殺害予告 文藝春秋が注意喚起
ITmedia NEWS / 2024年7月23日 17時10分
-
3シリーズ続編で「化けた」ゲームと言えば?
Game*Spark / 2024年7月23日 12時45分
-
4プロが本気で“アンパンマンの塗り絵”をしたら…… 衝撃の仕上がりが360万再生「凄すぎて笑うしかないww」「チーズが、、、」
ねとらぼ / 2024年7月18日 22時0分
-
5「画期的!」「すげえ」 テレ東が特番放送→新聞の番組表に描かれた“まさかのデザイン”に驚き
ねとらぼ / 2024年7月22日 19時26分
複数ページをまたぐ記事です
記事の最終ページでミッション達成してください