ヒト常在菌の個別解析、新時代へ
共同通信PRワイヤー / 2024年10月3日 12時0分
画像6
3万個の細菌ゲノム解読、抗生物質耐性遺伝子を追跡
2024年10月3日
早稲田大学
bitBiome株式会社
ヒト常在菌の個別解析、新時代へ 3万個の細菌ゲノム解読、抗生物質耐性遺伝子を追跡
詳細は早稲田大学HPをご覧ください。
発表のポイント
● がん・炎症性腸疾患などの患者と健常者を含む日本人被検者51名から、世界最大規模3万個のヒト常在菌※1のシングルセルゲノム解析※2を実施。
● 1.7万個の口腔内細菌・腸内細菌の高精度ゲノム情報を含むデータセットbbsag20を公開。
● 従来の手法(メタゲノム解析※3)では見落とされていた300種以上の腸内細菌のゲノムを取得。
● 遺伝子の「運び屋」可動性遺伝因子※4を介した抗生物質耐性遺伝子※5の広がりを個々の細菌レベルで解明。
● 細菌の抗生物質耐性の広がり方をより深く理解する手がかりを提供し、将来的な医療や公衆衛生への応用に期待。
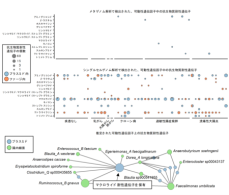
【画像:https://kyodonewsprwire.jp/img/202410027513-O2-d9heWb3Q】
図:本研究で用いた新しいシングルセルゲノム解析手法
私たちの健康に重要な役割を果たすヒト常在菌。しかし、その全容解明には個々の細菌を詳しく調べる必要があり、これまでの技術では困難でした。早稲田大学理工学術院の細川正人(ほそかわまさひと)准教授と早稲田大学発スタートアップbitBiome社の研究グループは、革新的なシングルセルゲノム解析技術を用いて、この課題に挑戦し、世界最大規模の3万個の口腔内細菌および腸内細菌の個別ゲノム解析を行いました。構築したbbsag20データセットからは、従来法では見落とされていた数百種の細菌のゲノム・遺伝子が発見されました。また、抗生物質耐性遺伝子やその「運び屋」の存在が個々の細菌単位で明らかになり、細菌間での遺伝子のやり取りを詳細に調査することが可能になりました。この成果は、常在菌を対象とした個別化医療や新たな抗生物質耐性対策の開発に貢献する可能性があります。
本研究成果は、2024年10月2日(水)(現地時間)にSpringer NatureグループのBioMed Central社が発刊するオープンアクセス科学誌「Microbiome」で公開されました。
論文名:A Single Amplified Genome Catalog Reveals the Dynamics of Mobilome and Resistome in the Human Microbiome
この記事に関連するニュース
-
前立腺がんの有無による腸内細菌の違いを発見 腸内細菌から前立腺がんのリスクを予測する手法の開発等に貢献
Digital PR Platform / 2024年9月26日 20時5分
-
阪大、薬に強い菌に対して解析し細菌の形態と薬剤耐性の関連を解明
マイナビニュース / 2024年9月20日 20時55分
-
中国が世界で最も整った海洋微生物遺伝子バンクを構築―中国メディア
Record China / 2024年9月11日 16時40分
-
マイクロバイオームに特化したヘルスケア企業KINS、本社およびラボを東京都江東区新木場に拡張移転
PR TIMES / 2024年9月11日 15時40分
-
京大、腸内細菌叢が幼児の気質と関連を持つことを発見
マイナビニュース / 2024年9月9日 20時40分
ランキング
-
1国慶節休暇前夜のテコ入れで中国株が急騰。「今度こそ」景気回復はあるか?
トウシル / 2024年10月3日 7時30分
-
2トヨタ、空飛ぶ車開発に追加出資 米新興企業ジョビーに730億円
共同通信 / 2024年10月3日 0時9分
-
3EUのエンジン車販売禁止計画、伊提案の見直し前倒しにドイツ反対
ロイター / 2024年10月3日 9時51分
-
4親が気づかない「できる子」の自己肯定感の低さ 手がかかる下の子ばかり気に掛けていたら…
東洋経済オンライン / 2024年10月3日 8時0分
-
5東証反発、一時1000円超高 首相・日銀総裁会談契機に
共同通信 / 2024年10月3日 12時16分
複数ページをまたぐ記事です
記事の最終ページでミッション達成してください